L’utilisation du NGS dans le diagnostic des infections sur prothèses d’épaule : une sensibilité meilleure que la culture est-elle mieux ?
Next-generation sequencing for diagnosis of infection: is more sensitive really better?
Allison J Rao, Ian S MacLean, Amanda J Naylor, Grant E Garrigues, Nikhil N Verma , Gregory P Nicholson
J Shoulder Elbow Surg. 2020 Jan;29(1):20-26.
Les auteurs s’interrogent sur l’opportunité et l’intérêt de la réalisation de séquençage haut débit lors de la prise en charge de prothèse articulaire d’épaule (chirurgie initiale). En d’autres termes, cette technologie a-t-elle sa place dans l’arsenal des techniques utilisées en diagnostic microbiologique ?
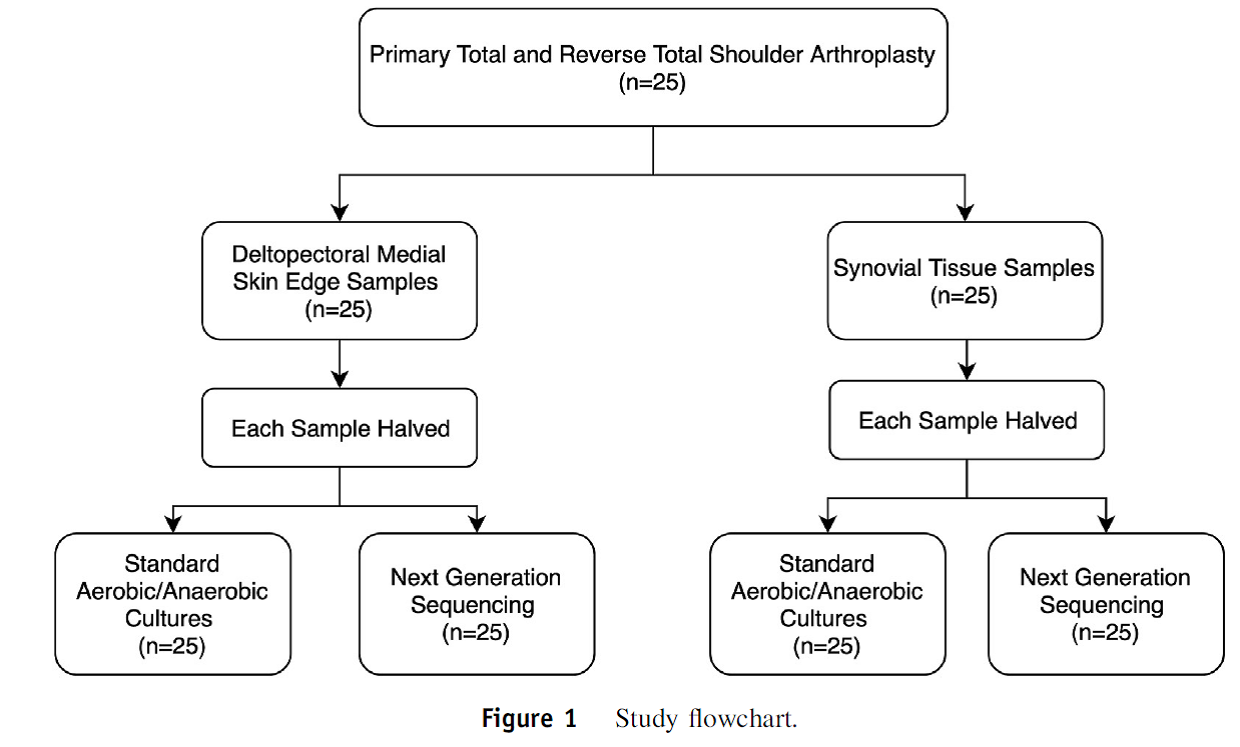
Méthodes. Etude prospective monocentrique, inclusion de 25 patients (17 prothèses inversées, 8 prothèses anatomiques). Comparaison de la culture après homogénéisation des prélèvements : 5 milieux solides aérobies incubés à 37°C en aérobiose pendant 14 jours et un milieu solide anaérobie incubé à 37°C en anaérobiose pendant 14 jours et du séquençage NGS sur une biopsie cutanée et un prélèvement profond prélevés lors de la pose de la prothèse et séparés en deux. Le séquençage NGS a été effectué via la technologie Ion Torrent PGM sequencer avec contrôle de qualité et détermination d’un seuil de prise en compte des séquences significatives.
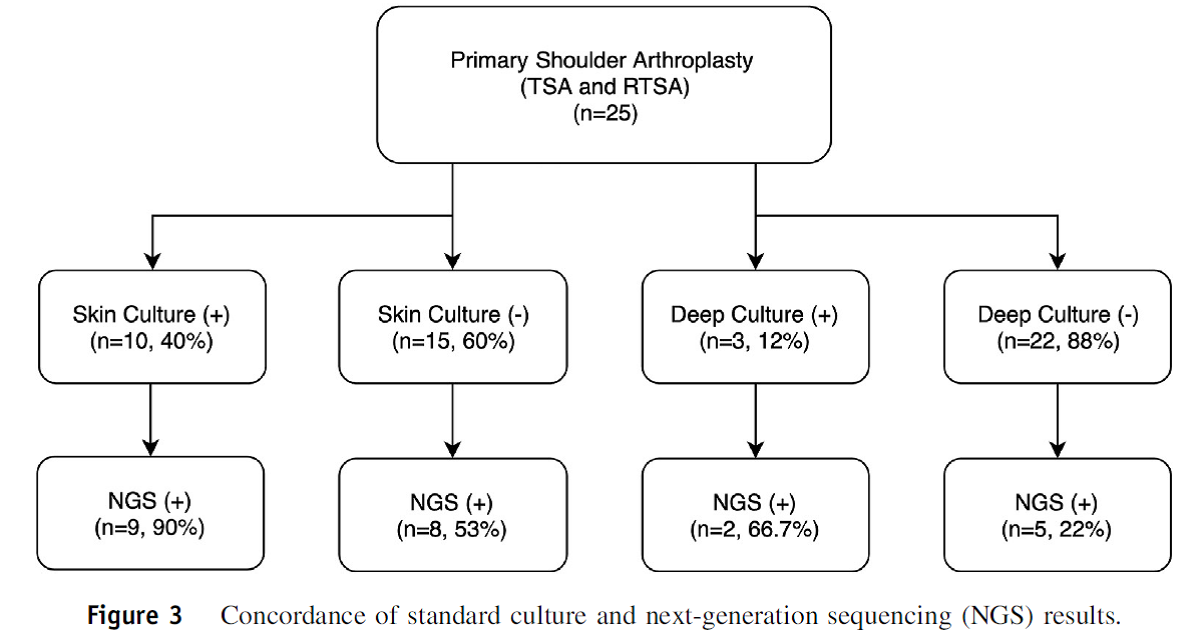
Résultats. Lors de ces chirurgies initiales de pose de prothèse, parmi les 25 prélèvements de biopsies cutanées, 10 étaient positifs en culture (40 %) vs 17 (68 %) en séquençage NGS. Parmi les 25 prélèvements de tissus profonds, 3 étaient positifs en culture (12 %) vs 7 (28 %) en séquençage NGS.
La concordance entre les cultures et le NGS a été analysée :
- Pour les 10 prélèvements superficiels positifs, huit étaient monomicrobiens en culture dont 7 positifs en NGS avec la même bactérie au seuil fixé de 10 %. Deux étaient positifs avec deux bactéries en culture. Le NGS retrouve les deux bactéries dans un cas et une seule bactérie dans l’autre cas.
- Pour les trois prélèvements profonds positifs, tous étaient monomicrobiens en culture et le NGS retrouve la même bactérie dans deux cas sur trois.
Concernant les espèces identifiées à partir des prélèvements superficiels, les plus fréquentes étaient C. acnes dans 44 %, S. epidermidis dans 24 % et E. coli dans 20 %. Pour les prélèvements profonds, l’ordre était inversé, E. coli dans 16 %, C. acnes dans 12 % et S. epidermidis dans 8 %.
D’autres bactéries étaient retrouvées par séquençage NGS, soit dans le prélèvement superficiel, soit dans le prélèvement profond, mais uniquement chez un patient.
Conclusion. L’application du séquençage NGS constitue une bonne approche pour l’identification d’infection, notamment polymicrobienne mais l’interprétation, les contraintes techniques, les seuils nécessitent de nouvelles études plus robustes en termes d’effectif et d’analyse. Les auteurs soulignent que sa plus grande sensibilité n’est peut-être pas un avantage.
Critique.
Cet article est intéressant car :
1/ Il soulève l’intérêt du NGS mais pose la question de son positionnement : intérêt dans l’épaule notamment dans la chirurgie initiale concourant à des confusions dans le texte et le titre (infection lors de la pose d’une prothèse ?) ? intérêt lors de la reprise ?
2/ Il démontre que cette approche n’est pas si simple d’interprétation, notamment au niveau de l’articulation de l’épaule pour laquelle l’antibioprophylaxie et la préparation cutanée ne suffisent pas à réduire les risques de sepsis ultérieurs, souvent torpides et insidieux de diagnostic parfois compliqué
3/ Points d’attention : Effectif petit donc prudence sur les % ; manque de précision quant à l’homogénéisation des prélèvements, absence de milieu liquide pour la culture, absence de flacon d’hémoculture ; manque de précisions méthodologiques notamment sur le seuil de 10 % en termes de charge bactérienne (nombre de reads ?) ou de bruit de fond potentiel ; taux de positivité en NGS à E. coli et culture négative (5 patients en superficiel et 4 en profond, est-ce les mêmes et rôle des réactifs de biologie moléculaire ? Etonnant les bactéries détectées en superficiel : Arthrobacter sp. (bactérie de l’environnement), Porphyromonas uenonis et Ureaplasma urealyticum (flore buccale et rhinopharynx = contaminants ?) ou en profond : Neisseria shayeganii (flore orale), Catenabacterium mitsuokai et Megamonas funiformis (flore fécale) !
Stéphane CORVEC, Bactériologie, CHU de Nantes